Cell Context
Défi : Accès à l’identité des cellules individuelles dans leur contexte spatial d’origine
Aperçu
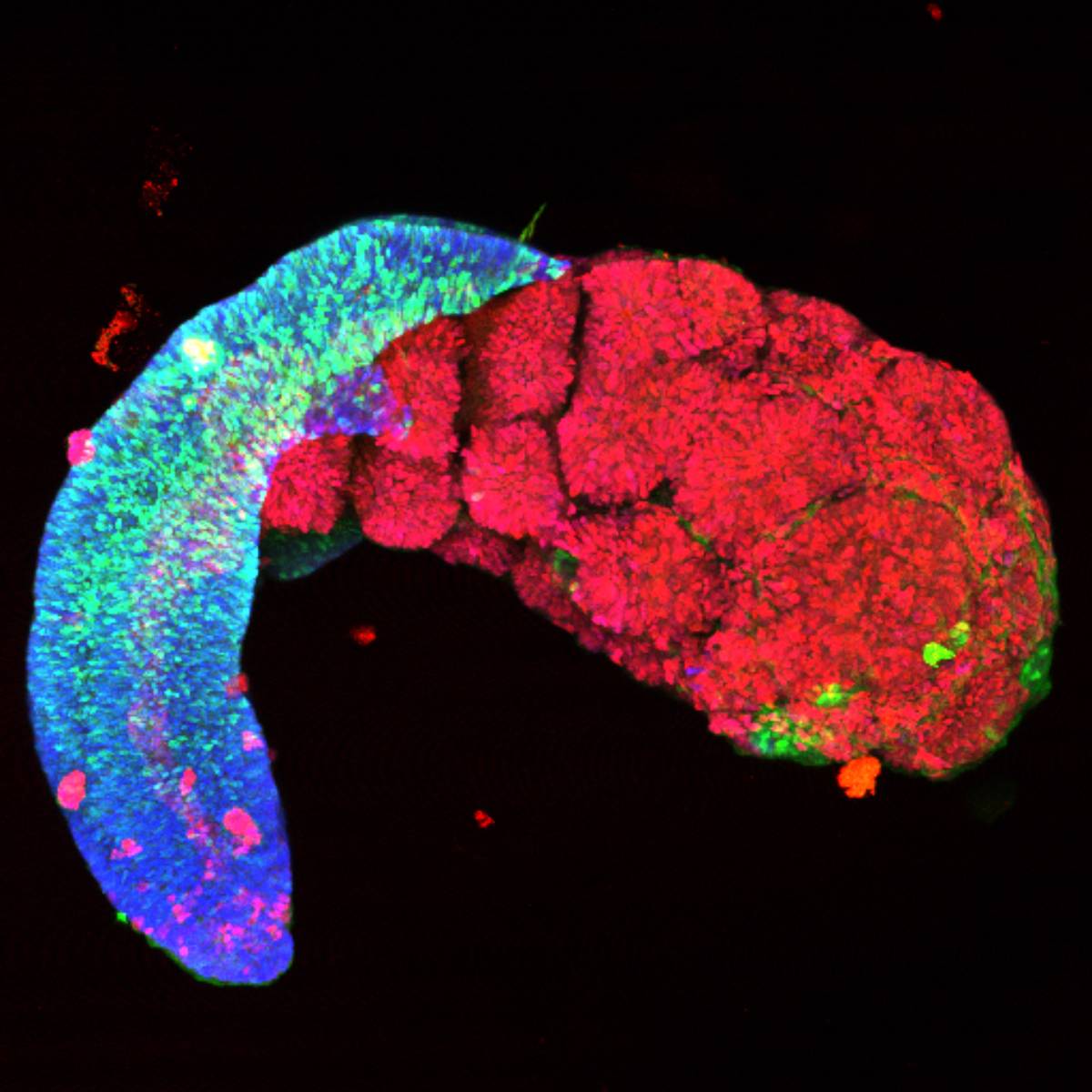
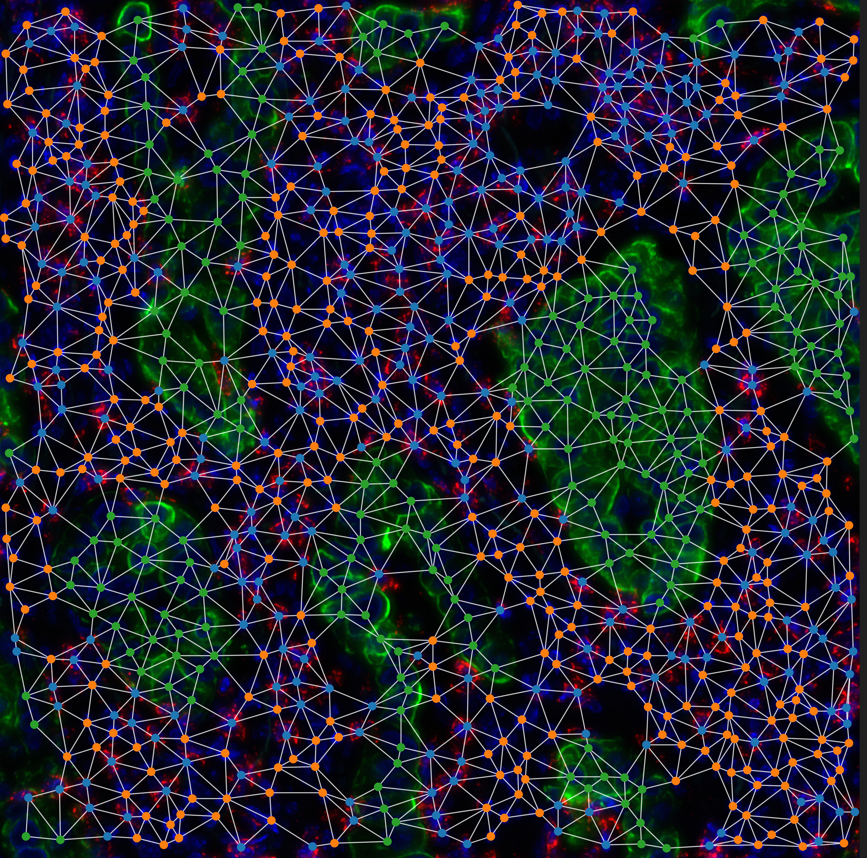
Le projet Cell Context développe des approches innovantes de multi-omique spatiale et unicellulaire pour étudier le développement neural et cérébral physiologique et une sélection de tumeurs pédiatriques du cerveau. Ces technologies permettent d’analyser, à l’échelle d’une seule cellule, les variations transcriptomiques, protéiques, épigénétiques et génétiques tout en maintenant l’organisation spatiale des tissus.
Giacomo Cavalli, responsable de l’équipe « 3D genome folding, function of Polycomb and Trithorax proteins in epigenetic heritance » de l’Institut de Génétique Humaine.
Marcelo Nollmann, responsable de l’équipe « Mécanismes de la ségrégation de l’ADN et du remodelage » du Centre de biologie structurale du CNRS et de l’Inserm.
Analyse des changements génomiques dans chaque cellule d’un tissu lors de la différentiation.

Objectifs clés
Développement de technologies de pointe
- Basée sur le séquençage
- Basée sur l’imagerie
- Basée sur la protéomique à cellule unique
Trois actions clés
Intégration de séquençage et de l’imagerie multi-omique à cellule unique appliquée au neurodéveloppement et au cancer pédiatrique :
- Séquençage des isoformes d’ARN.
- Analyse multi-omique du transcriptome, des marques épigénétiques et de l’accessibilité de la chromatine et de l’épigénome à l’échelle de cellules individuelles.
- Étude de l’intégrité du génome et de la transcription au niveau d’une cellule unique.
Imagerie spatiale Développements d’imageries multi-omic spatial adaptés au neurodéveloppement :
- Technologies de marquage, amplification des signaux, et clarification) pour l’imagerie multiplexée spatiale.
- Microscopie multiplexé adaptés aux échantillons épais
- Imagerie spatiale multimodale.
- Imagerie spatio-temporelle multidimensionnelle.
Protéomique pour le neurodéveloppement et le profilage des tumeurs cérébrales pédiatriques à l’échelle de la cellule unique :
- Utilisation des spectromètres de masse de nouvelle génération pour l’analyse du protéome à cellule unique.
- Développement et comparaison des approches complémentaires.
- Affinage de l’interprétation des données protéomiques avec les résultats transcriptomiques.
- Utilisation des pipelines expérimentaux et analytiques pour caractériser l’origine des tumeurs cérébrales pédiatriques.
Résultats attendus
- Progresser dans les techniques de multi-omiques à cellule unique et de séquençage
- Combinaison de la multi-omique à cellule unique avec le séquençage long-read pour la caractérisation des isoformes
- Multiplexage de l’épigénomique en cellule unique pour des biopsies congelées et des modèles afin de cartographier l’évolution épigénomique des tumeurs
- Multi-omique en cellule unique pour cartographier les paysages de dommages à l’ADN en relation avec la transcription
- Améliorer les technologies de génomique spatiale et d’imagerie
- Méthodes d’imagerie transcriptomique Hi-M/in situ pour les informations spatiales en 3D sur l’ARN et les omiques de la chromatine
- Imagerie hyperspectrale multicolore rapide pour caractériser les états cellulaires et les signatures de la chromatine
- Nouvelles modalités d’imagerie à cellule unique (vélocité de l’ARN, imagerie des mutations ponctuelles, imagerie de traçage des lignées par code-barres)
- Combiner Méthodes d’imagerie transcriptomique Hi-M/in situ pour les récupérer des informations spatiales multi-omiques en 3D
- Imagerie hyperspectrale multicolore rapide pour caractériser les états cellulaires et les états épigénétiques.
- Combinaison de l’imagerie de cellules vivantes avec Hi-M/in situ transcriptomique pour corréler les lignées ou les phénotypes des cellules individuelles avec leurs états transcriptomiques spécifiques
- Progresser dans les méthodes de protéomique à cellule unique
- Méthodes de protéomique à cellule unique.
- Pipeline bioinformatique pour la protéomique à cellule unique.
- Identifier les dérèglements de lignée et la progression tumorale en utilisant la protéomique à cellule unique dans les tumeurs cérébrales pédiatriques.
Ce projet contribuera au développement des techniques multi-omiques qui pourront être utilisées afin de mieux comprendre les mécanismes du neurodéveloppement humain et aux changements qui ont lieu dans les modèles des cancers pédiatriques.
Les autres projets PEPR