DATA MED
Projet Data : Exploiter la puissance des données pour comprendre les cancers pédiatriques
Aperçu
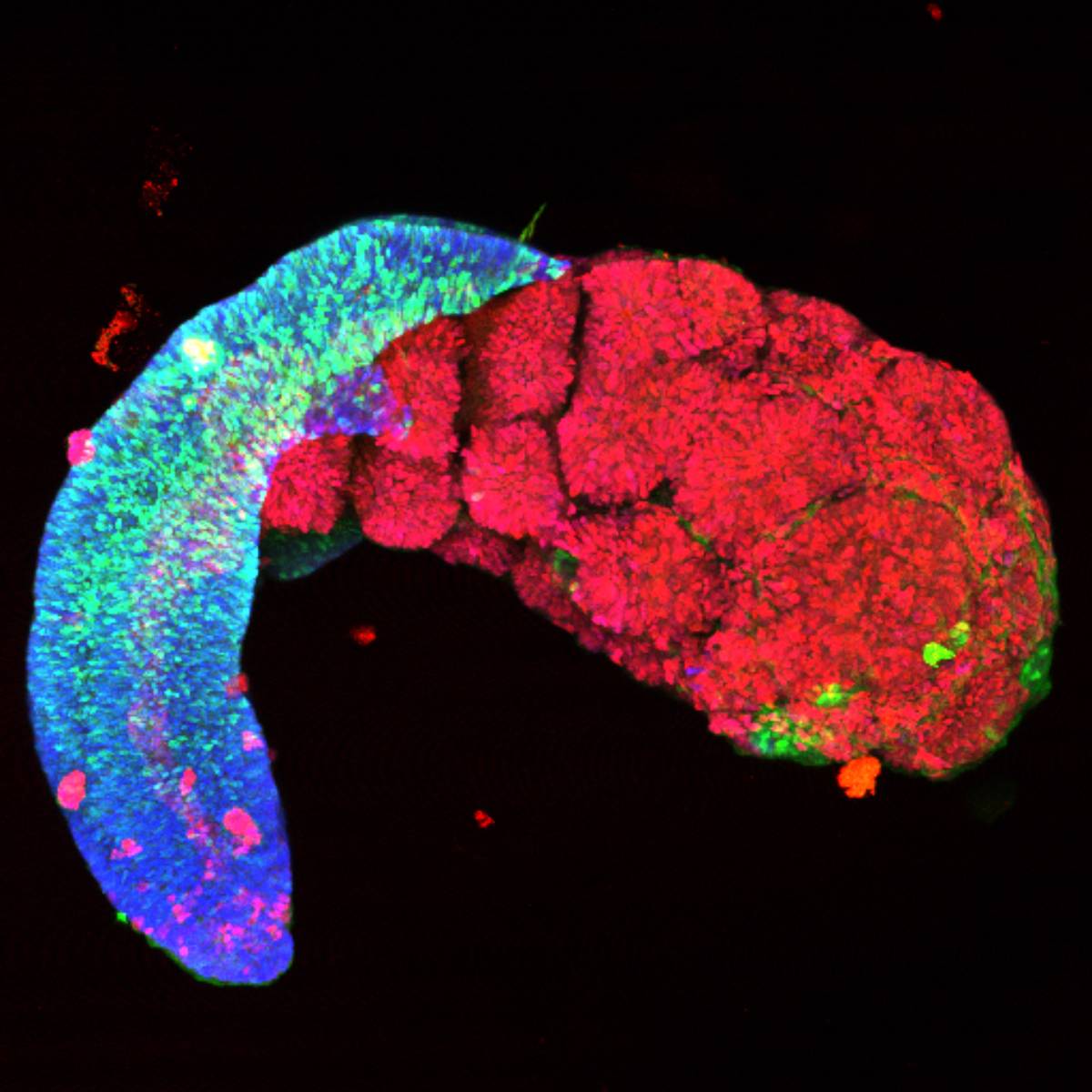
Le projet Data de Cell-ID répond aux défis posés par l’analyse des données massives et complexes issues de technologies innovantes. Ces données, générées à l’échelle unicellulaire, sont essentielles pour décrypter les mécanismes qui dérèglent le destin cellulaire et provoquent le développement de cancers pédiatriques.
Thomas Walter, responsable de l’équipe « Statistical Machine Learning and Modelling of Biological Systems » de l’unité U1331 « Computational Oncology » de l’Institut Curie.
Daniel Jost, responsable de l’équipe « Biologie physique de la chromatine » de l’unité UMR 5239 et INSERM 1293 « Biologie et modélisation de la cellule » (LBMC).

Objectifs clés
- Créer une infrastructure de gestion des données capable de stocker, partager et sécuriser les données tout en garantissant leur conformité aux contraintes légales et éthiques.
- Développer des outils d’analyse basés sur l’intelligence artificielle pour extraire des informations cruciales à partir de données omiques, d’images et d’autres sources.
- Assurer l’interopérabilité avec les initiatives internationales afin de maximiser l’impact des découvertes et d’intégrer les données à des projets de grande envergure.
Moyens déployés
Infrastructure de gestion des données
- Mise en place d’un plan de gestion des données (DMP) pour définir les règles de collecte, de documentation, de sécurité et de partage.
- Déploiement d’une cyberinfrastructure capable de traiter des données à l’échelle du pétaoctet, en s’appuyant sur des réseaux locaux et nationaux, comme la plateforme de bioinformatique de l’Institut Curie.
- Développement d’un portail web sécurisé permettant un accès centralisé aux données anonymisées et prétraitées pour l’analyse en aval.
Outils d’analyse et d’intégration
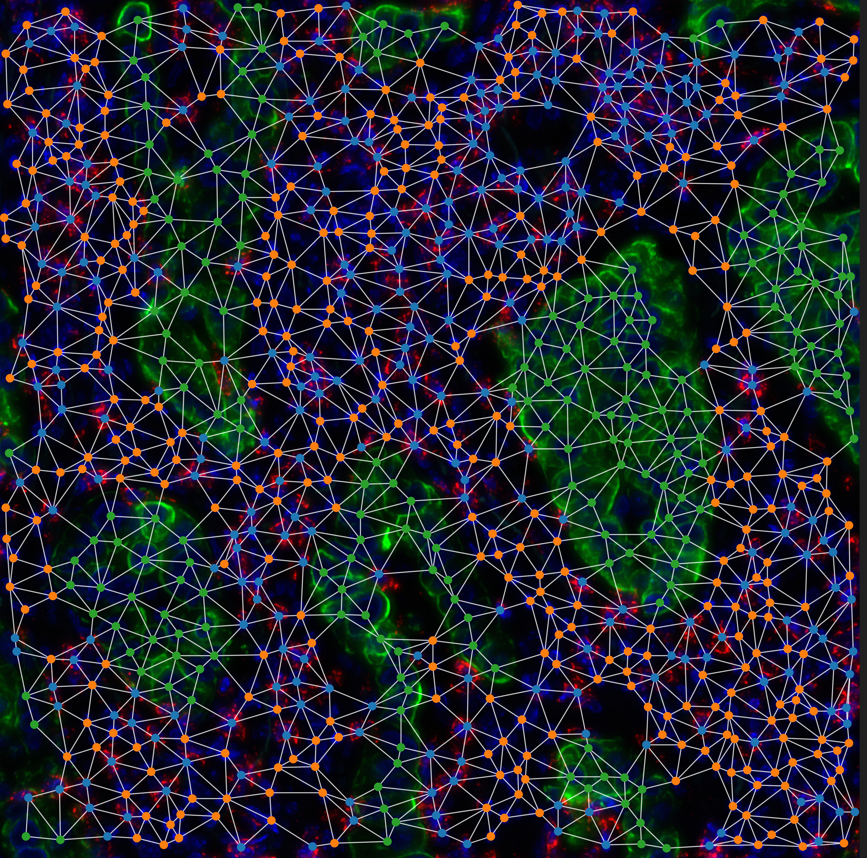
- Création de pipelines bioinformatiques pour prétraiter et analyser les données issues de technologies comme la transcriptomique spatiale ou la spectrométrie de masse.
- Conception de modèles d’intelligence artificielle capables d’imputer des données manquantes, d’analyser des images en 2D et 3D, et de relier les informations omiques aux phénotypes cellulaires.
- Développement de solutions d’intégration multimodale pour combiner différentes sources de données et reconstruire les trajectoires cellulaires à travers le temps et l’espace.
Partage et diffusion des résultats
- Publication des données et des outils développés en open access, en suivant les principes FAIR (Facile à trouver, Accessible, Interopérable, Réutilisable).
- Collaboration avec des initiatives internationales telles que le Human Cell Atlas (HCA) et Euro-BioImaging pour assurer la visibilité et l’interopérabilité des données produites.
Résultats attendus
Le projet Data offrira une ressource inédite pour la recherche sur les cancers pédiatriques, comprenant :
Une base de données centralisée des profils cellulaires.
- Des pipelines et algorithmes accessibles à la communauté scientifique.
- Des modèles prédictifs permettant d’identifier les anomalies précoces et de proposer des stratégies d’interception thérapeutique.
En combinant infrastructures, IA et collaborations internationales, le projet Data de Cell-ID vise à transformer les données en connaissances exploitables pour améliorer la prise en charge des cancers pédiatriques.
Projet Med: Intercepter les tumeurs cérébrales pédiatriques
Aperçu
Le projet MED vise à comprendre et à intercepter l’apparition et la progression de tumeurs cérébrales pédiatriques, telles que :
- Médulloblastome (MB)
- Gliomes pédiatriques de haut grade (pHGG), incluant :
- Gliomes diffus de la ligne médiane (DMG)
- Gliomes hémisphériques (DHG) avec mutations de l’histone H3.3
- Tumeurs rhabdoïdes tératoïdes atypiques (ATRT)
Laure Bally-Cuif, responsable de l’équipe « Neurogénétique du poisson zébré» de l’unité CNRS UMR3778 « Biologie du développement et cellules souches » de l’Institut Pasteur.
David Castel chercheur dans l’équipe « Génomique et oncogénèse des tumeurs cérébrales pédiatriques » de l’UMR 981 « Prédicteurs moléculaires et nouvelles cibles en oncologie » de l’Institut Gustave Roussy.
Pourquoi ces tumeurs ?
- Complexité de la « cellule d’origine » : une même mutation peut générer des tumeurs différentes selon le type cellulaire d’origine ou le microenvironnement.
- Altérations épigénétiques communes : ces tumeurs présentent fréquemment des mutations touchant la régulation de la chromatine.
- Développement précoce : ces cancers se développent souvent dès la période embryonnaire ou peu après la naissance.
Objectifs clés
Cartographier les étapes précoces du développement tumoral
- Étudier les signatures (épi)génétiques et les cellules d’origine dans des modèles animaux (souris, drosophile) et des organoïdes cérébraux.
- Identifier les événements qui dérèglent la différenciation cellulaire et favorisent la transformation tumorale.
Valider les hypothèses dans des modèles humains
- Utiliser des organoïdes cérébraux dérivés de cellules souches de patients pour recréer et étudier le développement des tumeurs.
- Exploiter des échantillons de tumeurs primaires pour identifier des signatures spécifiques de dérèglement cellulaire.
Identifier des « second hits » favorisant la progression tumorale
- Réaliser des criblages CRISPR pour découvrir des mutations ou altérations épigénétiques critiques.
- Tester des traitements ciblant ces altérations dans les modèles expérimentaux.
Traduire les découvertes en interventions cliniques
- Développer des stratégies pour détecter et traiter les récidives tumorales.
- Tester des « épidrogues » et d’autres agents thérapeutiques visant à restaurer la différenciation cellulaire ou à ralentir la progression tumorale.
Impact à long terme
Les résultats du projet MED permettront :
- Un diagnostic plus précoce grâce à l’identification de signatures moléculaires spécifiques.
- De nouvelles approches thérapeutiques pour améliorer le suivi et la prise en charge des tumeurs cérébrales pédiatriques.
- Des applications élargies pour d’autres types de cancers pédiatriques et des troubles neurodéveloppementaux liés.
Les autres projets PEPR